
CHIP 是一种用于在体内条件下高效捕获蛋白质与 DNA 特定区域相互作用的强大工具。其本质是一个 “在细胞内进行的抗原 - 抗体反应”,允许我们回答一个关键问题:“在某个特定时刻,我感兴趣的蛋白质(转录因子、组蛋白修饰等)结合在基因组的哪些位置上?”
整个过程遵循一个清晰的逻辑链,其核心步骤可用以下流程图概括:
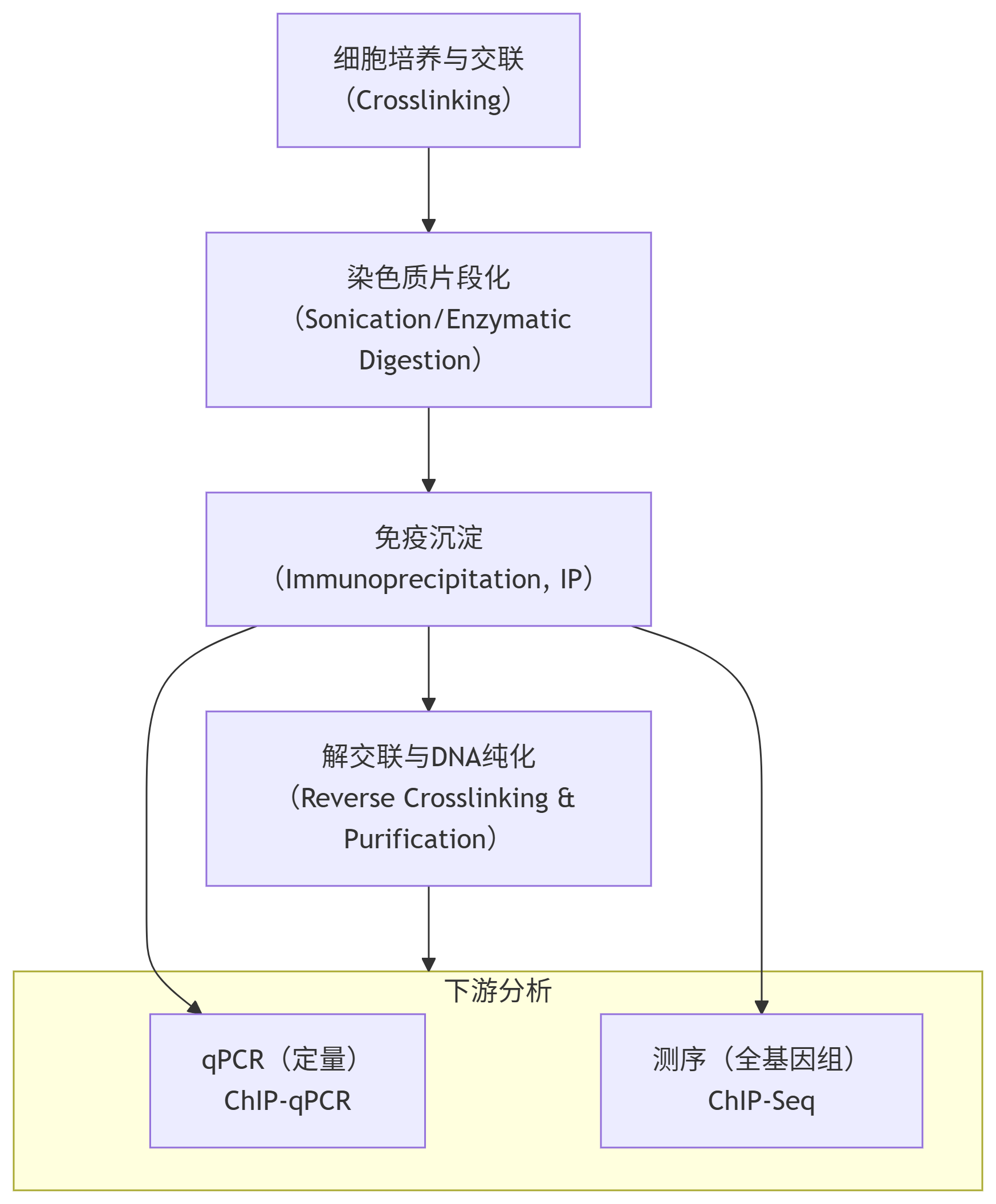
生物学意义:每一步都至关重要。交联固定了瞬时的蛋白 - DNA 相互作用;片段化决定了分辨率的极限;IP 的特异性是整个实验成败的关键;下游分析决定了信息的深度和广度。
失败的 CHIP 实验浪费的不仅是时间,更是珍贵的样本。以下是在设计阶段必须深思熟虑的要点:
这是 CHIP 实验的 “阿喀琉斯之踵”。
• 首选:CHIP 级(ChIP-grade)抗体。供应商通常会提供相关验证数据。
• 验证:如果没有 CHIP 级抗体,必须进行验证。
◦ 阳性对照:使用已知表达目标蛋白和结合位点的细胞系。
◦ 阴性对照:使用不表达该蛋白的细胞系或使用 IgG 同型抗体。
◦ 敲低 / 敲除验证(黄金标准):在 KO 或 KD 细胞系中进行 CHIP,信号应显著减弱或消失。
• 甲醛(Formaldehyde):最常用,适用于大多数转录因子和组蛋白修饰。交联时间(通常 5-30 分钟)需要优化,过长会导致抗原表位遮蔽和染色质难以片段化。
• 双交联:对于结合不稳定的蛋白,可先用 DSG(Disuccinimidyl glutarate)进行蛋白 - 蛋白交联,再用甲醛进行蛋白 - DNA 交联。
• 天然 CHIP(Native CHIP):不使用交联剂,仅用于研究组蛋白修饰(如 H3K27me3, H3K4me3 等),因其相互作用非常稳定。优点是背景低、信噪比高。
• 超声处理(Sonication):最普遍的方法。目标是获得200-1000 bp的片段(理想大小是 200-500 bp)。关键点:
◦ 优化!优化!再优化!不同细胞类型、数量、仪器所需条件截然不同。
◦ 保持低温,防止交联逆转。
◦ 通过琼脂糖凝胶电泳或 Bioanalyzer 检测片段化效率。
• 酶解法(Micrococcal Nuclease, MNase):主要用于 Native CHIP。它酶切核小体连接区,产生单核小体或寡核小体水平的片段,分辨率更高。
一个严谨的 CHIP 实验必须包含以下对照:
• Input DNA:保留一部分片段化后的染色质(占 IP 反应的 1%-10%),不进行 IP,直接解交联纯化。这是你的 “总 DNA” 对照,用于校正 PCR/qPCR 效率和在 ChIP-seq 中构建基线。
• 阴性 IgG 抗体:非免疫宿主的 IgG,用于评估非特异性结合背景。
• 阳性对照抗体:针对一种已知广泛存在的组蛋白修饰(如 H3K4me3 于活跃启动子,H3K27me3 于抑制区域),验证整个实验体系有效。
• 阴性对照引物:针对已知不结合目标蛋白的基因组区域设计 qPCR 引物。
1. 交联:用 1% 甲醛处理细胞 10-15 分钟(室温),后用甘氨酸淬灭。
2. 细胞裂解:用含蛋白酶抑制剂的裂解液裂解细胞,获取细胞核。
3. 染色质片段化:超声处理至所需大小,离心取上清。
4. 免疫沉淀:
◦ 取部分染色质作为 Input 保存。
◦ 剩余部分与特异性抗体和 Protein A/G 磁珠在 4°C 下孵育过夜。
1. 洗脱与解交联:用低 pH 洗脱缓冲液将结合复合物从磁珠上洗下,加入 NaCl(至终浓度 200mM),65°C 加热过夜以逆转交联。
2. DNA 纯化:使用蛋白酶 K 消化蛋白,随后用酚氯抽提或商业试剂盒纯化 DNA。
3. 下游分析:
• 目的:验证特定候选区域的富集情况。
• 数据分析 - 富集倍数(Fold Enrichment)计算:
◦ % Input 法:%Input = 2^[Ct(Input) - Ct(IP)] * F * 100%
(F 为 Input 稀释因子,如 Input 占 IP 的 10%,则 F=10)。
◦ 相对富集法(ΔΔCt 法):将 IP 样本相对于 Input 的 Ct 值,与阴性对照区域或 IgG 对照的 Ct 值进行比较。Fold Enrichment = 2^[-(ΔCt(IP) - ΔCt(neg))]
• 目的:在全基因组范围内发现所有结合位点。
• 数据分析流程:
a. 质控与比对:FastQC 检查数据质量,使用 Bowtie2/BWA 等工具将 reads 比对到参考基因组。
b. 峰检测(Peak Calling):使用 MACS2 等算法,比较 IP 样本与 Input 对照,识别出显著富集的基因组区域(称为 “Peaks”)。
c. 注释与可视化:将 Peaks 注释到最近的基因启动子、增强子等元件上。使用 IGV 等基因组浏览器直观查看。
d. Motif 分析:在 Peak 序列中寻找富集的 DNA 序列 motif,可推断结合蛋白的合作因子或识别其共识序列。
e. 差异结合分析:比较不同条件下(如处理 vs 对照)的 ChIP-seq 数据,找出结合发生显著变化的区域。
问题 | 可能原因 | 解决方案 |
低信号 / 无富集 | 抗体效率低或失效;交联不足;片段化过度 / 不足 | 验证抗体;优化交联和时间;检查染色质片段大小 |
高背景 | 抗体非特异性结合;洗涤不充分 | 使用预清除步骤;增加洗涤严格性(提高盐浓度);确保抗体特异性 |
Input 或 IP DNA 降解 | 操作过程中被核酸酶污染 | 使用新鲜抑制剂;确保所有试剂无菌 |
ChIP-seq 背景噪音高 | 细胞数量不足;测序深度不够;Input 对照质量差 | 确保足够细胞起始量(通常 > 10^6);增加测序深度;制备高质量的 Input 库 |
qPCR 重复性差 | 技术操作误差;DNA 纯化后残留盐分或蛋白 | 规范操作;对纯化后的 DNA 进行二次沉淀或柱纯化 |
• CUT&Tag/RUN:新型的体内染色质分析技术,利用融合蛋白(Protein A-Tn5 转座酶)靶向目的蛋白,直接在细胞内进行标签化和测序文库构建。优势:信噪比极高、所需细胞数极少(可单细胞)、背景低、流程快。
• Multi-omics 整合:将 ChIP-seq 数据与 ATAC-seq(开放染色质)、RNA-seq(基因表达)、Hi-C(三维基因组)等数据进行整合分析,构建多维度的基因调控网络。
• 单细胞 ChIP-seq(scChIP-seq):尽管技术挑战巨大,但仍是理解细胞异质性的终极方向。
CHIP 技术自问世以来,一直是解析基因调控奥秘的基石。它既是一门科学,也是一门艺术。成功的 CHIP 实验源于严谨的设计、经过验证的试剂、优化的流程和恰当的数据分析。希望这篇指南能为您扫清迷雾,助您的研究之路更加顺畅。
记住,在分子生物学中,控制好你的对照,你就控制了一切。皮诺飞生物专注生命科学研究技术服务!更多请咨询工作人员!